[논문리뷰] - U-Net : Convolutional Networks for Biomedical Image Segmentation, MICCAI 2015
이미지로부터 객체(Object)를 추출하는 것은 컴퓨터 비전 분야에서 매우 중요한 Task 중 하나입니다. 예를들어 의료분야에서 종양 및 세포를 구분하고 표시하는 것은 의사들이 병을 진단을 할 때 큰 도움을 줄 수 있습니다. 또한 자율주행과 같이 Task에서는 물체를 구분하는 기능이 선 수행되어야 가능합니다.
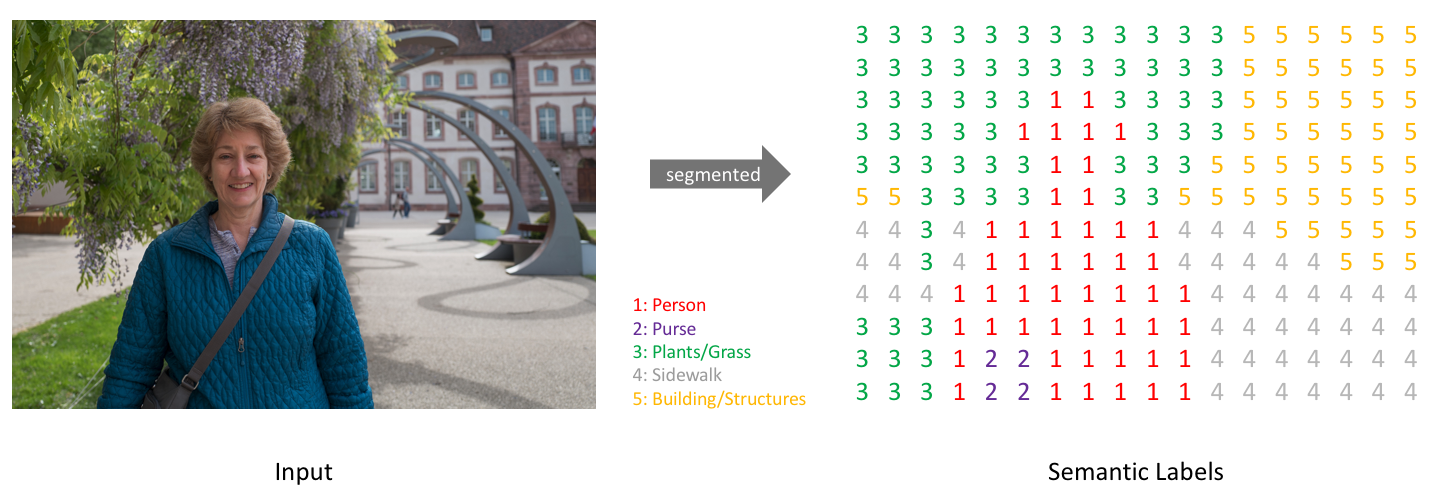
오늘 포스팅은 객체를 인식하고 분류하는 다양한 방법 중에서 픽셀 기반으로 이미지를 분할하여 구분하는 모델인 U-net or UNet 에 대해 상세하게 리뷰하도록 하겠습니다.
이 글은 U-net 논문과 MEDIUM 을 참고하여 정리하였음을 먼저 밝힙니다.
논문 그대로를 리뷰하기보다는 생각을 정리하는 목적으로 제작하고 있기 때문에 실제 내용과 다른점이 존재할 수 있습니다.
혹시 제가 잘못 알고 있는 점이나 보안할 점이 있다면 댓글 부탁드립니다.
Short Summary
이 논문의 큰 특징 2가지는 아래와 같습니다.
- 넓은 범위의 이미지 픽셀로부터 의미정보를 추출하고 의미정보를 기반으로 각 픽셀마다 객체를 분류하는 U 모양의 아키텍처를 제시합니다.
- 서로 근접한 객체 경계를 잘 구분하도록 학습하기 위하여 Weighted Loss를 제시합니다.
모델 구조
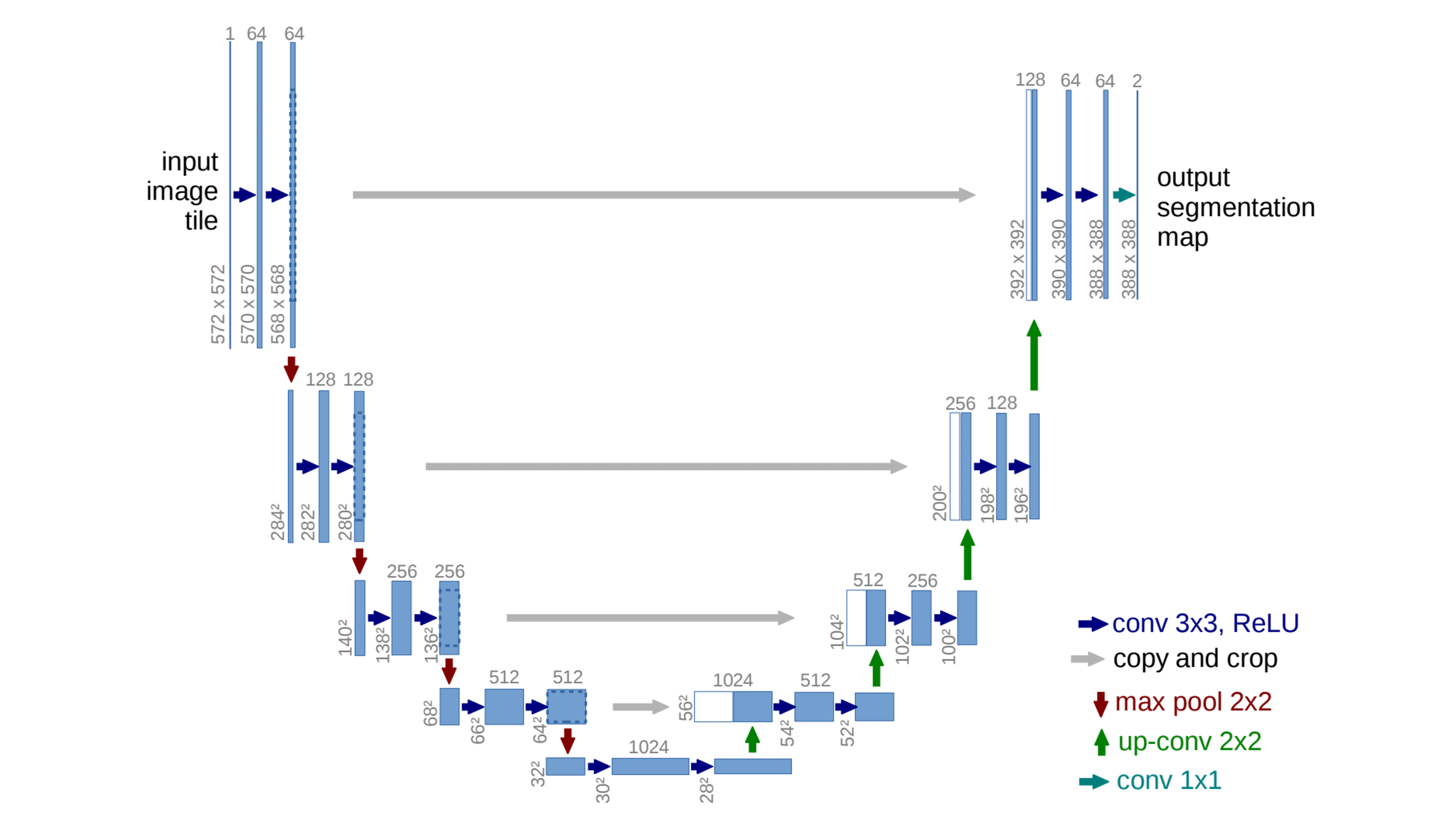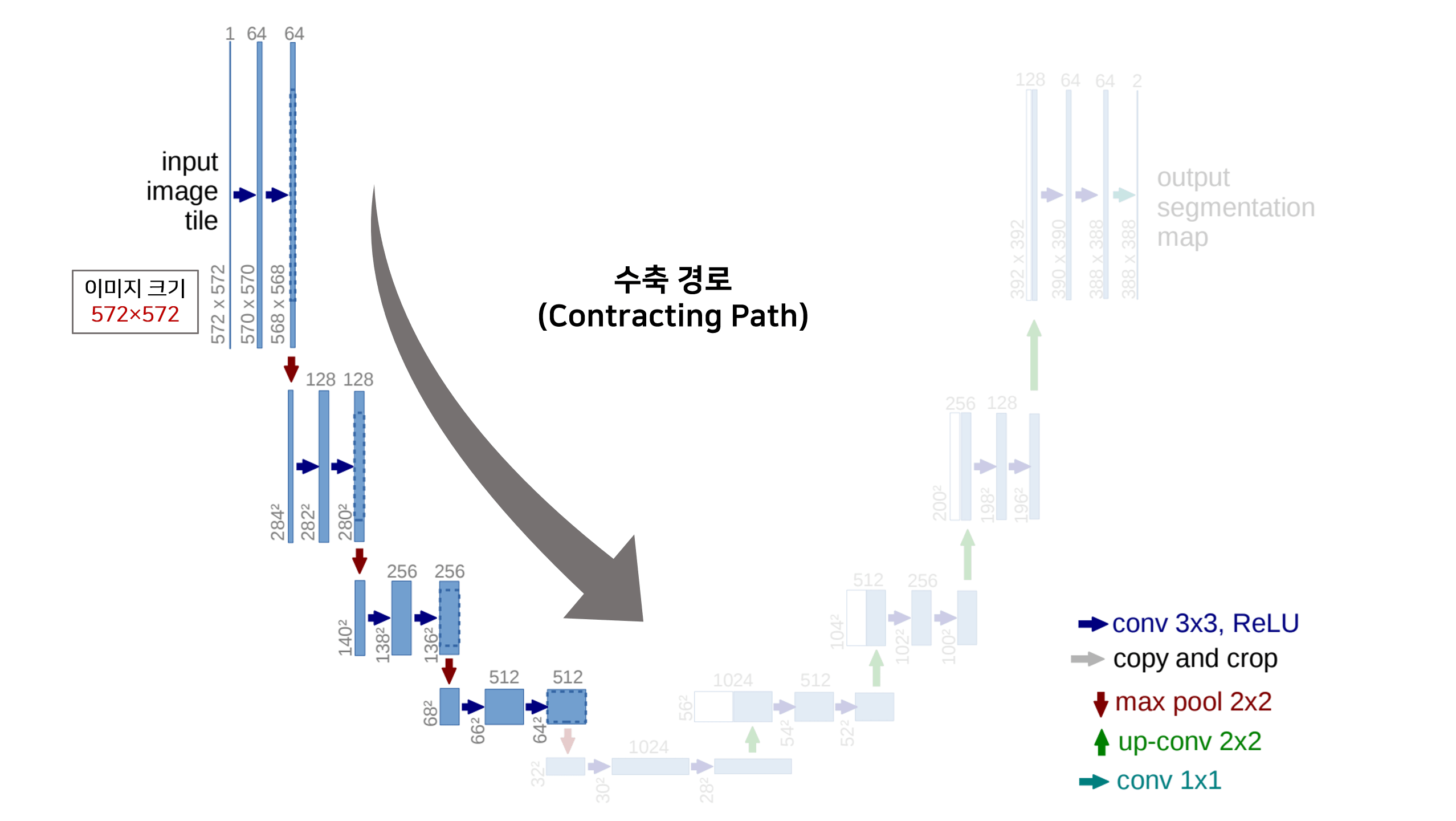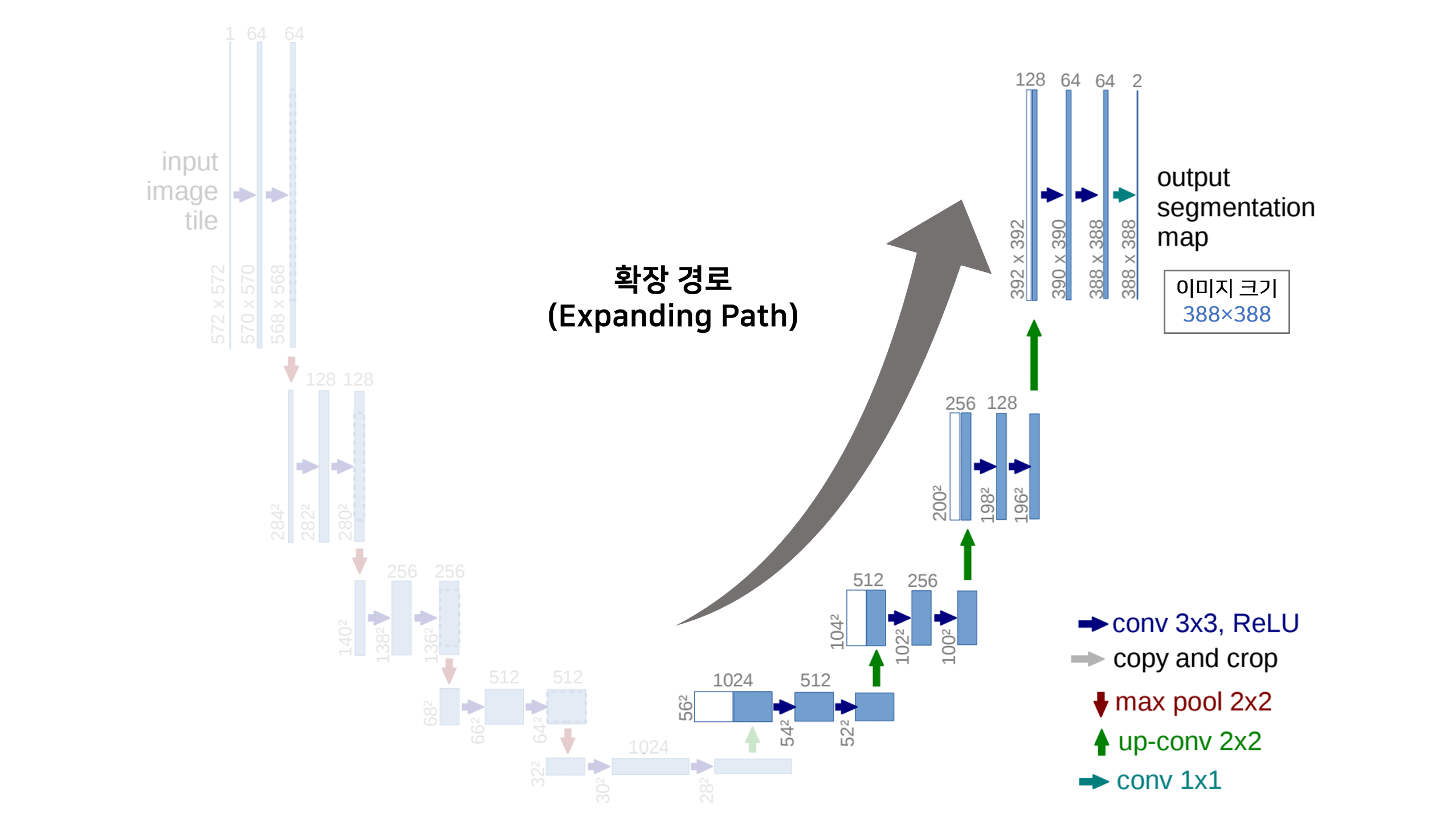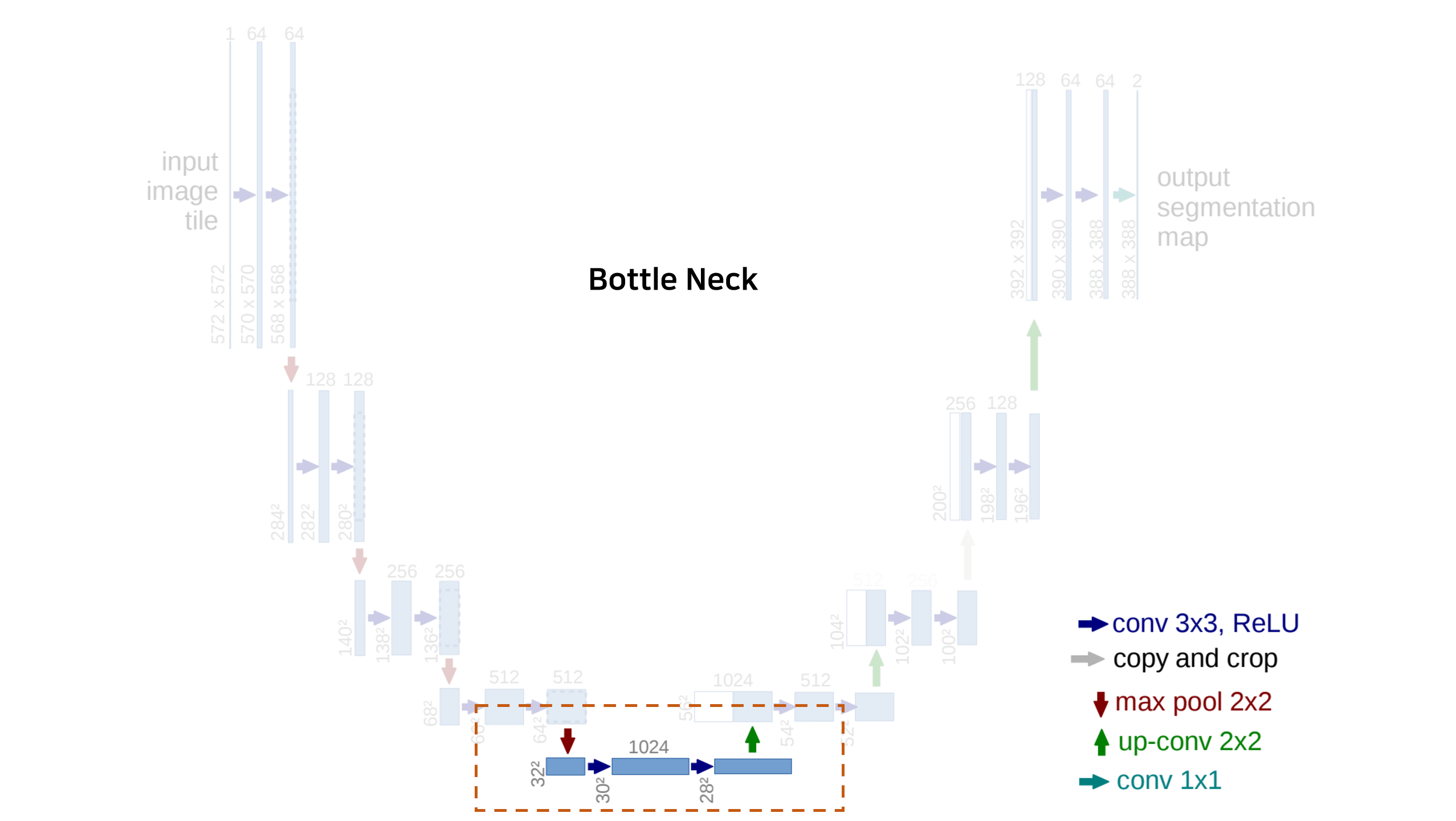
U를 닮은 모델의 구조는 크게 3가지로 나눌 수 있습니다.
- 점진적으로 넓은 범위의 이미지 픽셀을 보며 의미정보(Context Information)을 추출하는 수축 경로(Contracting Path)
- 의미정보를 픽셀 위치정보와 결합(Localization)하여 각 픽셀마다 어떤 객체에 속하는지를 구분하는 확장 경로(Expanding Path)
- 수축 경로에서 확장 경로로 전환되는 전환 구간(Bottle Neck)
모델의 Input은 이미지의 픽셀별 RGB 데이터이고 모델의 Output은 이미지의 각 픽셀별 객체 구분 정보(Class)입니다.
Convolution 연산과정에서 패딩을 사용하지 않으므로 모델의 Output Size는 Input Size보다 작습니다.
예를들어 572×572×(RGB) 크기의 이미지를 Input으로 사용하면 Output으로 388×388×(Class) 이미지가 생성됩니다.
$Input(Width × Height × RGB) -> Model -> Output(Width × Height × Class)$
1) 수축 경로(Contracting Path)
수축 경로에서 아래와 같은 Downsampling 과정을 반복하여 특징맵(Feature Map)을 생성합니다.
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1)
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1)
- 2×2 Max-polling Layer (Stride 2)
수축경로는 주변 픽셀들을 참조하는 범위를 넓혀가며 이미지로부터 Contextual 정보를 추출하는 역할을 합니다. 3×3 Convolution을 수행할 때 패딩을 하지 않으므로 특징맵(Feature Map)의 크기가 감소합니다. Downsampling 할 때 마다 채널(Channel)의 수를 2배 증가시키면서 진행합니다. 즉 처음 Input Channel(1)을 64개로 증가시키는 부분을 제외하면 채널은 $1->64->128->256->512->1024$ 개로 Downsampling 진행할 때마다 증가합니다.
논문에서는 Batch-Normalization이 언급되지 않았으나 구현체 및 다수의 리뷰에서 Batch-Normalization을 사용하는 것을 확인하였습니다. [참고자료]
2) 전환 구간(Bottle Neck)
수축 경로에서 확장 경로로 전환되는 구간입니다.
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1)
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1)
- Dropout Layer
마지막에 적용된 Dropout Layer는 모델을 일반화하고 노이즈에 견고하게(Robust) 만드는 장치입니다.
3) 확장 경로(Expanding Path)
확장 경로에서 아래와 같은 Upsampling 과정을 반복하여 특징맵(Feature Map)을 생성합니다.
- 2×2 Deconvolution layer (Stride 2)
- 수축 경로에서 동일한 Level의 특징맵(Feature Map)을 추출하고 크기를 맞추기 위하여 자른 후(Cropping) 이전 Layer에서 생성된 특징맵(Feature Map)과 연결(Concatenation)합니다.
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1)
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1)
확장경로는 2)Skip Connection을 통해 수축 경로에서 생성된 Contextual 정보와 위치정보 결합하는 역할을 합니다. 동일한 Level에서 수축경로의 특징맵과 확장경로의 특징맵의 크기가 다른 이유는 여러번의 패딩이 없는 3×3 Convolution Layer를 지나면서 특징맵의 크기가 줄어들기 때문입니다. 확장경로의 마지막에 Class의 갯수만큼 필터를 갖고 있는 1×1 Convolution Layer가 있습니다. 1×1 Convolution Layer를 통과한 후 각 픽셀이 어떤 Class에 해당하는지에 대한 정보를 나타내는 3차원(Width × Height × Class) 벡터가 생성됩니다.
학습 방법
본 논문에서 다양한 학습 장치들을 통해 모델의 성능을 향상시킵니다.
- Overlap-tile strategy : 큰 이미지를 겹치는 부분이 있도록 일정크기로 나누고 모델의 Input으로 활용합니다.
- Mirroring Extrapolate : 이미지의 경계(Border)부분을 거울이 반사된 것처럼 확장하여 Input으로 활용합니다.
- Weight Loss : 모델이 객체간 경계를 구분할 수 있도록 Weight Loss를 구성하고 학습합니다.
- Data Augmentation : 적은 데이터로 모델을 잘 학습할 수 있도록 데이터 증강 방법을 활용합니다.
Overlap-tile strategy
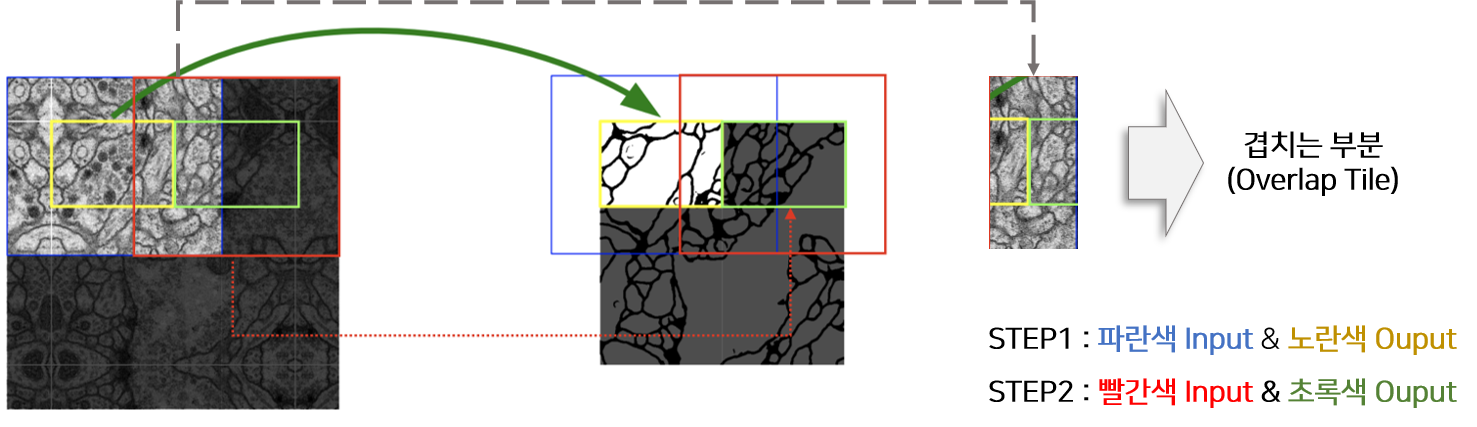
위 그림은 MEDIUM BLOG 에서 만든 그림을 참고하여 재구성 하였습니다. 이미지의 크기가 큰 경우 이미지를 자른 후 각 이미지에 해당하는 Segmentation을 진행해야 합니다. U-Net은 Input과 Output의 이미지 크기가 다르기 때문에 위 그림에서 처럼 파란색 영역을 Input으로 넣으면 노란색 영역이 Output으로 추출됩니다. 동일하게 초록색 영역을 Segmentation하기 위해서는 빨간색 영역을 모델의 Input으로 사용해야 합니다. 즉 겹치는 부분이 존재하도록 이미지를 자르고 Segmentation하기 때문에 Overlap Tile 전략이라고 논문에서는 지칭합니다.
Mirroring Extrapolate
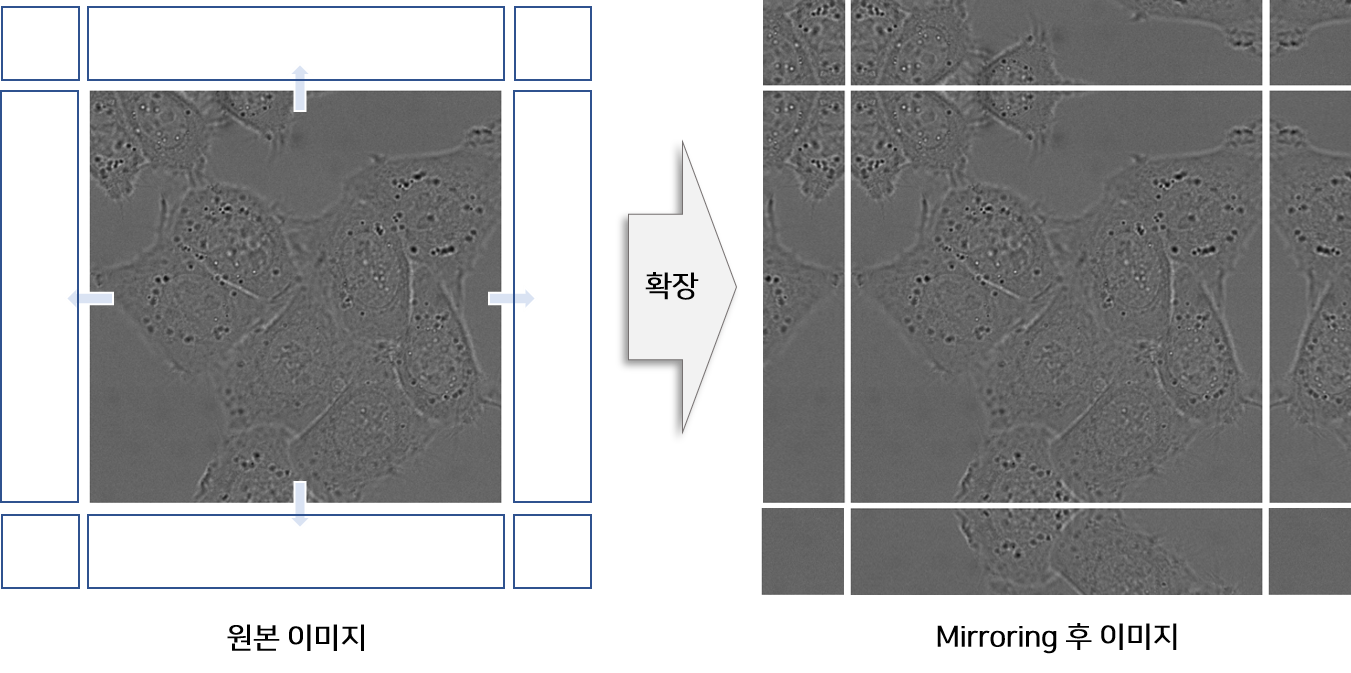
이미지의 경계부분을 예측할 때에는 Padding을 넣어 활용하는 경우가 일반적입니다. 본 논문에서는 이미지 경계에 위치한 이미지를 복사하고 좌우 반전을 통해 Mirror 이미지를 생성한 후 원본 이미지의 주변에 붙여 Input으로 사용합니다.
본 논문의 실험분야인 biomedical 에서는 세포가 주로 등장하고, 세포는 상하 좌우 대칭구도를 이루는 경우가 많기 때문에 Mirroring 전략을 사용했을 것이라고 추측합니다.
Weight Loss

모델은 위 그림처럼 작은 경계를 분리할 수 있도록 학습되어야 합니다. 따라서 논문에서는 각 픽셀이 경계와 얼마나 가까운지에 따른 Weight-Map을 만들고 학습할 때 경계에 가까운 픽셀의 Loss를 Weight-Map에 비례하게 증가 시킴으로써 경계를 잘 학습하도록 설계하였습니다.
$a_k(x)$ : 픽셀 x가 Class k일 값(픽셀 별 모델의 Output)
$p_k(x)$ : 픽셀 x가 Class k일 확률(0~1)
$l(x)$ : 픽셀 x의 실제 Label
$w_0$ : 논문의 Weight hyper-parameter, 논문에서 10으로 설정
$\sigma$ : 논문의 Weight hyper-parameter, 논문에서 5로 설정
$d_1(x)$ : 픽셀 x의 위치로부터 가장 가까운 경계와 거리
$d_2(x)$ : 픽셀 x의 위치로부터 두번째로 가까운 경계와 거리
$w(x)$는 픽셀 x와 경계의 거리가 가까우면 큰 값을 갖게 되므로 해당 픽셀의 Loss 비중이 커지게 됩니다. 즉 학습 시 경계에 해당하는 픽셀을 잘 학습하게 됩니다.
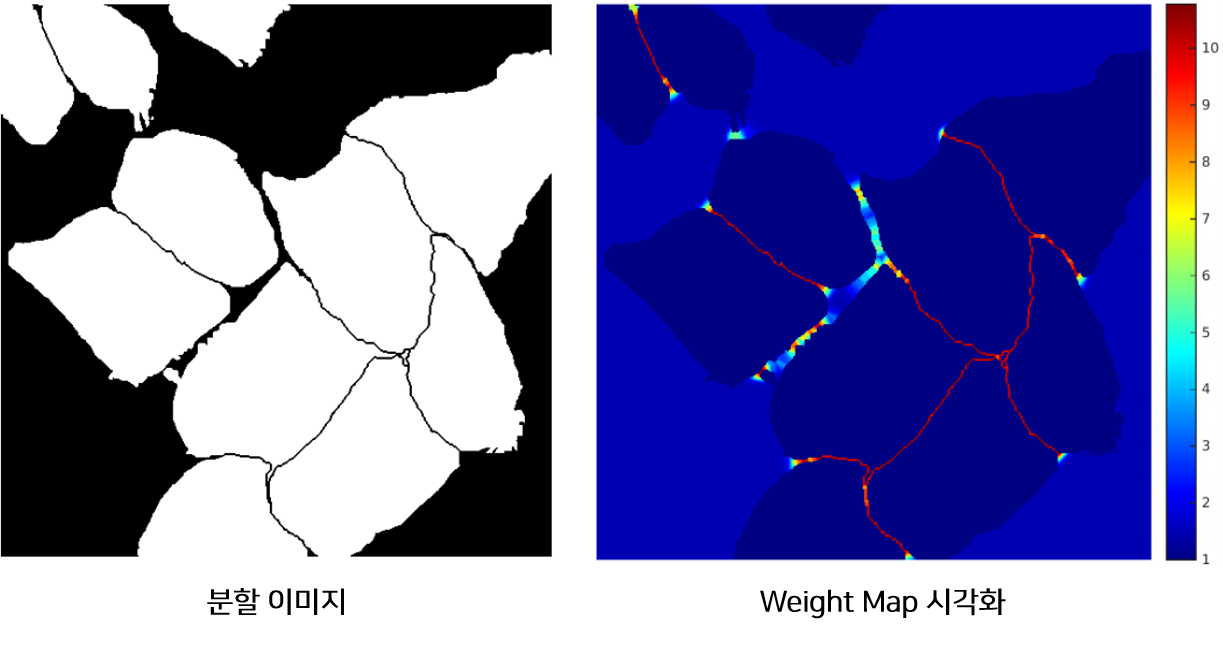
위 그림은 이미지의 픽셀 위치에 따른 Weight $w(x)$를 시각화한 것입니다. $w(x)$는 객체의 경계 부분에서 큰 값을 갖는 것을 확인할 수 있습니다.
객체간 경계가 전체 픽셀에 차지하는 비중은 매우 작습니다. 따라서 Weight Loss를 이용하지 않을 경우 경계가 잘 학습되지 않아 여러개의 객체가 한개의 객체로 표시 될 가능성이 높아 보입니다.
Data Augmentation
데이터의 양이 적기 때문에 데이터 증강을 통해 모델이 Noise에 강건하도록 학습시킵니다. 데이터 증강 방법으로 Rotation(회전), Shift(이동), Elastic distortion 등이 있습니다.
본 논문에서는 자세하게 데이터 증강방법을 묘사하지 않습니다. 이미지를 활용하여 데이터 증강하는 방법은 [Elastic distortion] 에서 참고바랍니다.
Others
- Gaussian Distribution 을 이용하여 모델의 파라미터를 초기화하고 학습합니다.
- 이미지를 최대한 크게 사용하고 Optimizer에 Momentum(0.99)를 부여하여 일관적이게 학습하도록 조절합니다.
실험 및 결과
모델의 성능을 평가하기 위하여 EM Segmentation challenge의 Dataset을 활용합니다. EM Segmentation challenge Dataset은 30개의 Training 데이터를 제공합니다. 각 데이터는 이미지와 함께 객체와 배경이 구분된(0 or 1) Ground Truth Segmentation Map을 포함하고 있습니다.
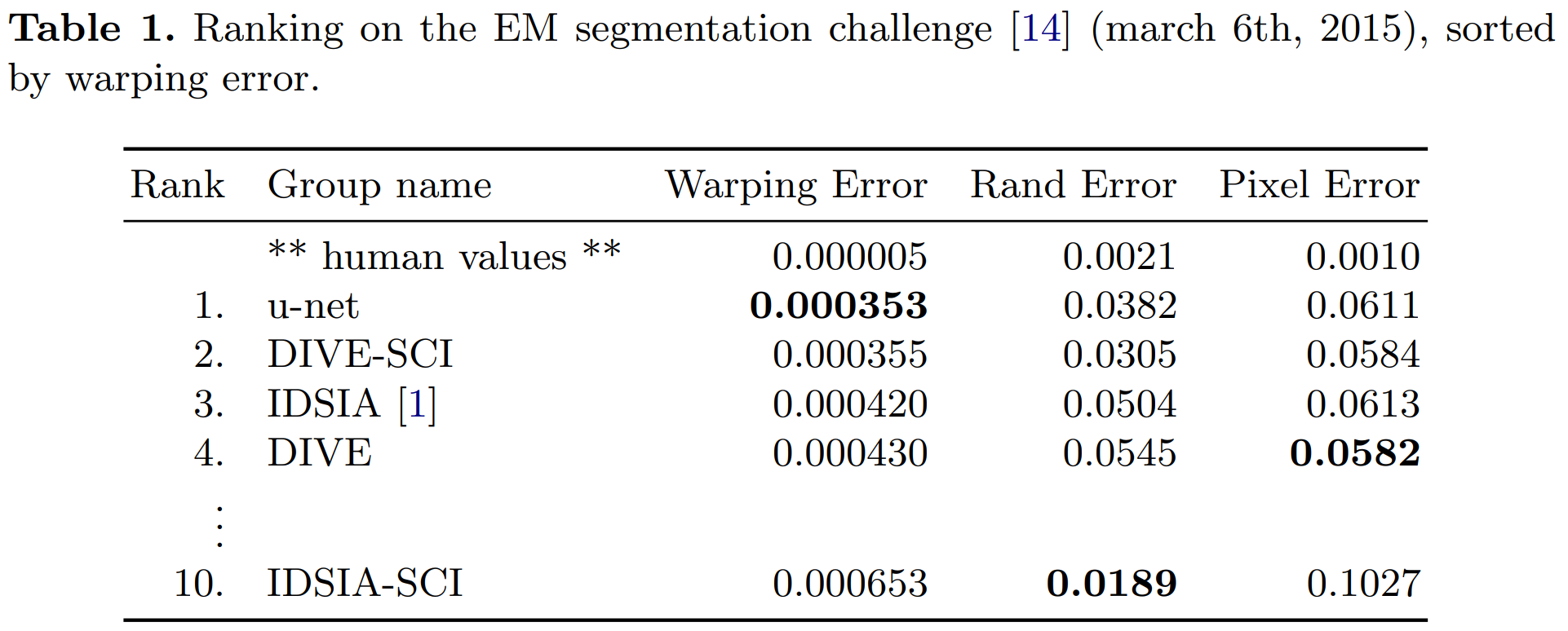
Warping Error : 객체 분할 및 병합이 잘 되었는지 세크멘테이션과 관련된 에러
Warping Error를 기준으로 “EM Segmentation” 데이터에서 U-Net 모델이 가장 좋은 성능을 보이고 있습니다.
세포 분류 대회인 ISBI cell tracking challeng 에서 모델의 성능을 평가한 표는 아래와 같습니다. “PhC-U373” 데이터는 위상차 현미경으로 기록한 35개의 이미지를 Training 데이터로 제공합니다. “DIC-HeLa” 데이터는 HeLa 세포를 현미경을 통해 기록하고 20개의 이미지를 Training 데이터로 제공합니다.
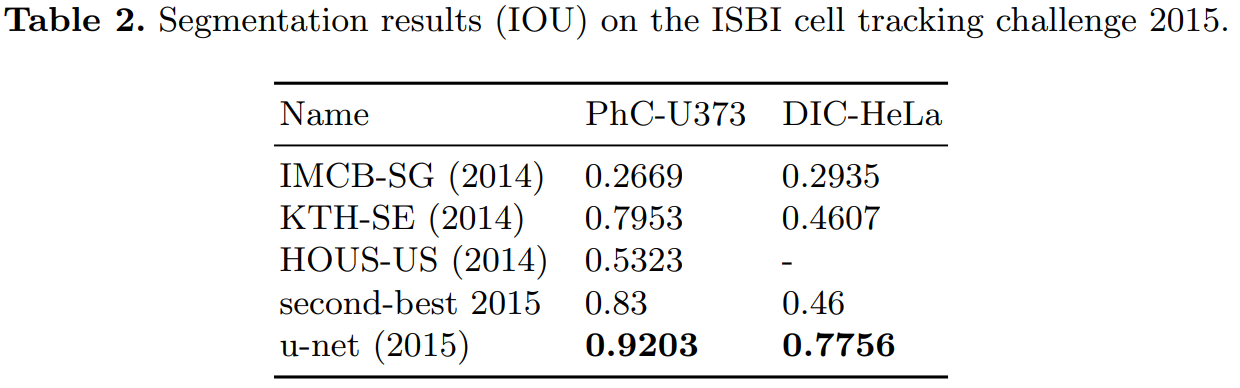
U-Net 모델은 “PhC-U373” 데이터에서 92% IOU Score를 획득하였으며 2등 모델이 획득한 점수 83% 와 현격한 차이를 보이고 있습니다. U-Net 모델은 “DIC-HeLa” 데이터에서 77.5% IOU Score를 획득하였으며 2등 모델이 획득한 점수 46% 와 현격한 차이를 보이고 있습니다.
결론 및 개인적인 생각
매우 효과적이고 실용적인 논문입니다. Skip Architecture는 Layer를 깊게 쌓을수 있게 하여 복잡한 Task를 잘 수행할 수 있게 합니다. 또한 Bottle Neck에서 생기는 정보의 손실을 줄이는 역할을 합니다. Weighted Loss는 근거리에 있는 객체를 효과적으로 분리하여 학습하는 좋은 방법입니다. 모델의 구조가 간단하여 구현이 쉽고 다양한 구현체 및 튜토리얼을 웹에서 검색할 수 있어 활용하기 좋습니다.
최근에 Kaggle 대회에서 U-Net 모델이 활용되어 좋은 점수를 받은 것을 보았습니다. 다양한 구조의 모델들이 고안되고 있지만 지금도 U-Net 모델의 구조는 Image Segmentation에 가장 효과적인 모델 중 하나인 것 같습니다.
Reference
- [BLOG] Semantic Segmentation 목적
- [BLOG]U-Net 논문 리뷰 — U-Net: Convolutional Networks for Biomedical Image Segmentation, 강준영
- [BLOG] U-Net Tutorial
- [BLOG] Elastic distortion, HJ harry
- [PAPER] U-Net: Convolutional Networks for Biomedical Image Segmentation(2015), Olaf Ronneberger
- [GITHUB] UNet: semantic segmentation with PyTorch